Aperçu du package
2024-03-19
mapMCDA_French.Rmd## Loading required package: sf## Linking to GEOS 3.10.2, GDAL 3.4.1, PROJ 8.2.1; sf_use_s2() is TRUE## Loading required package: terra## terra 1.7.65## Loading required package: mapMCDAIntroduction
Le package mapMCDA facilite la ponderation de plusieurs
facteurs de risque pour produîre une carte de risque
épidémiologique.
Néanmoins, l’expertise de l’utilisateur est cruciale. Elle s’exprime à trois niveaux :
Choix des facteurs de risque rélevants
Pour chaque facteur, mise en échelle commune de risque (e.g. entre 0 et 100)
Évaluation deux-à-deux de l’importance rélative des facteurs de risque
À continuation, on utilise des cartes fournis par le package pour produîre une carte de risque d’exemple. Cette carte n’a aucun valeur épidémiologique.
1. Facteurs de risque
La fonction mapMCDA_datasets() permet de charger en
mémoire toutes les cartes disponibles dans le package.
L’objet cmr est une simple liste de cartes : objets de
type sf pour les cartes vectoriels et de type
SpatRaster pour les cartes raster.
Pour utiliser d’autre cartographie, utiliser les fonctions
sf::st_read et terra::rast() respectivement
pour des cartes vectoriels ou raster.
On peut aussi bénéficier de la fonction load_layer()
pour charger tout type des cartes automatiquement, comme démontré dans
le code commenté.
cmr <- mapMCDA_datasets()
# layers <- list.files(
# system.file("cartography/CMR", package = "mapMCDA"),
# full.names = TRUE
# )
# cmr <- lapply(layers, load_layer)
# names(cmr) <- rmext(basename(layers)))L’une de ces cartes est celle des unités épidémiologiques, utilisée pour établir le cadre de travail.
unites_epi <- cmr$cmr_admin3
par(mar = c(0, 0, 0, 0))
plot(st_geometry(unites_epi))
Unités épidémiologiques d’exemple pour le Cameroun.
2. Mise en échelle
Chaque facteur de risque varie dans une échelle qui lui est propre. La densité animale, par exemple, varie entre 0 et presque 5500 têtes par \(km^2\).
Pour les cartes vectorielles, qui representent la localisation des entités spatiales telles que lacs ou forêts, on considère la distance à dites entités.
Ce package utilise pour l’instant une fonction linèaire pour la mise en échelle. Cependant, la rélation peut être directe ou inverse.
plot(
data.frame(x = c(0, 100), y = c(0, 100)),
type = 'l',
xaxs = "i",
yaxs = "i",
xaxt = "n",
lab = c(1, 1, 7),
xlab = c("Échelle originale"),
ylab = c("Échelle de risque")
)
abline(100, -1)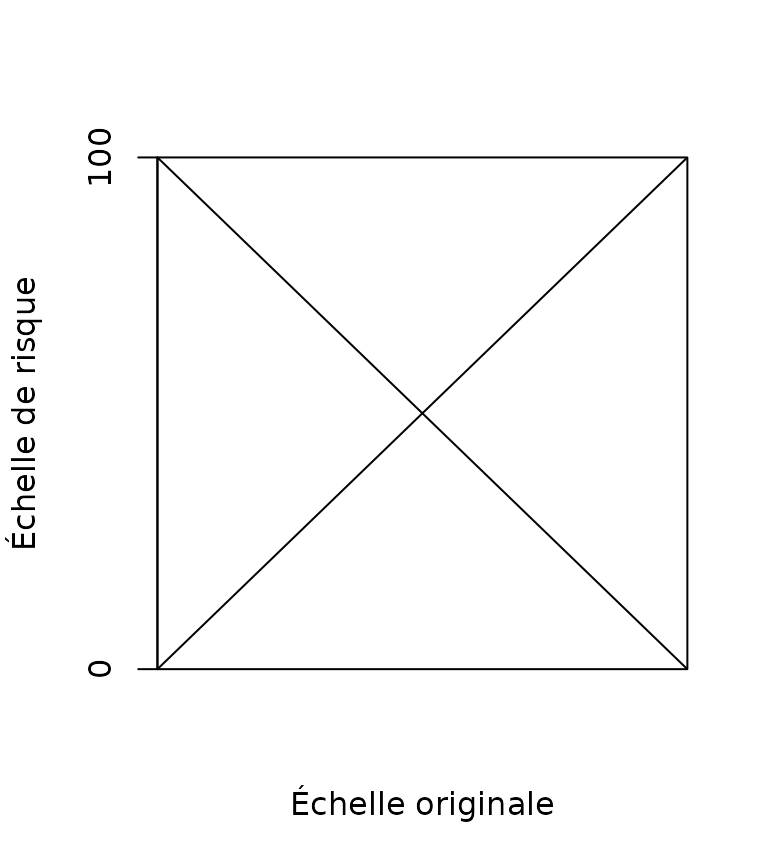
Mis en échelle directe ou inverse.
La fonction risk_layer() calcule la carte de risque
associé à un facteur concret dans une échelle donnée. Par défault c’est
entre 0 et 100. Pour inverser la rélation il suffit de passer les limits
dans l’ordre inverse, e.g. c(100, 0).
D’ailleurs, elle utilise la carte d’unités épidémiologiques pour établir les limits de calcul de risque.
risques <- list(
dens_animale = risk_layer(
cmr$animal.density,
boundaries = unites_epi
# , scale_target = c(0, 100) # échelle directe par défault
),
points_eau = risk_layer(
cmr$water_bodies,
boundaries = unites_epi,
scale_target = c(100, 0) # échelle renversée
),
parcs = risk_layer(
cmr$national_parks,
boundaries = unites_epi,
scale_target = c(100, 0) # échelle renversée
)
)##
|---------|---------|---------|---------|
=========================================
|---------|---------|---------|---------|
=========================================
On voudrait examiner les cartes de risque ainsi calculées. Mais pour cela, il faut qu’elles soient alignées. C’est à dire, qu’elles aient les mêmes extents, résolutions et projections.
On peut se servir de la fonction align_layers() qui
arrange tout ça pour nous. Noter que ce pas n’est pas nécessaire pour
continuer, car cette fonction est automatiquement executée si
besoin.
risques_alignes <- align_layers(risques)
plot(terra::rast(risques_alignes), nc = 3, col = grDevices::hcl.colors(50))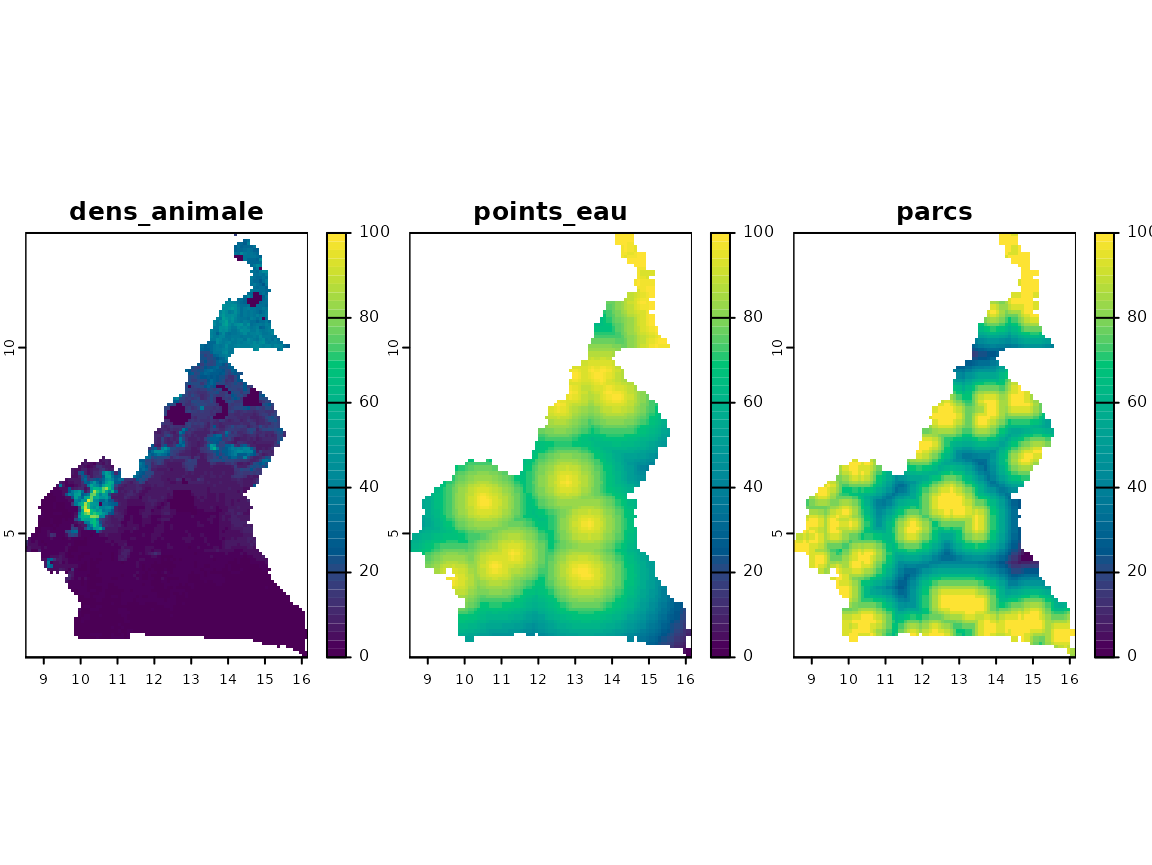
Niveaux de risque associé à chaque facteur.
3. Pondération des facteurs de risque
Il y a 3 facteurs à considérer. Il faut comparer 2-à-2 leurs importances relatives en une échelle de 1 à 9 et representer ces rélations en une matrice qui doit avoir des 1 dans son diagonale.
Noter que les élements symmetriques doivent être réciproques.
M <- matrix(c(
1, 6, 4,
1/6, 1, 3,
1/4, 1/3, 1
), byrow = TRUE, 3, 3)
colnames(M) <- rownames(M) <- names(risques)
knitr::kable(M, digits = 2)| dens_animale | points_eau | parcs | |
|---|---|---|---|
| dens_animale | 1.00 | 6.00 | 4 |
| points_eau | 0.17 | 1.00 | 3 |
| parcs | 0.25 | 0.33 | 1 |
Dans cette exemple, on considère que la densité animale est 6 fois plus importante que la distance aux points d’eau, et 4 fois plus importante que la distance aux parcs. Au même temps, que la distance aux points d’eau est 3 fois plus importante que celle aux parcs.
Le système calcule les coefficients de pondération les plus
consistents avec ces valorations par paires, avec la fonction
compute_weights().
w <- compute_weights(M)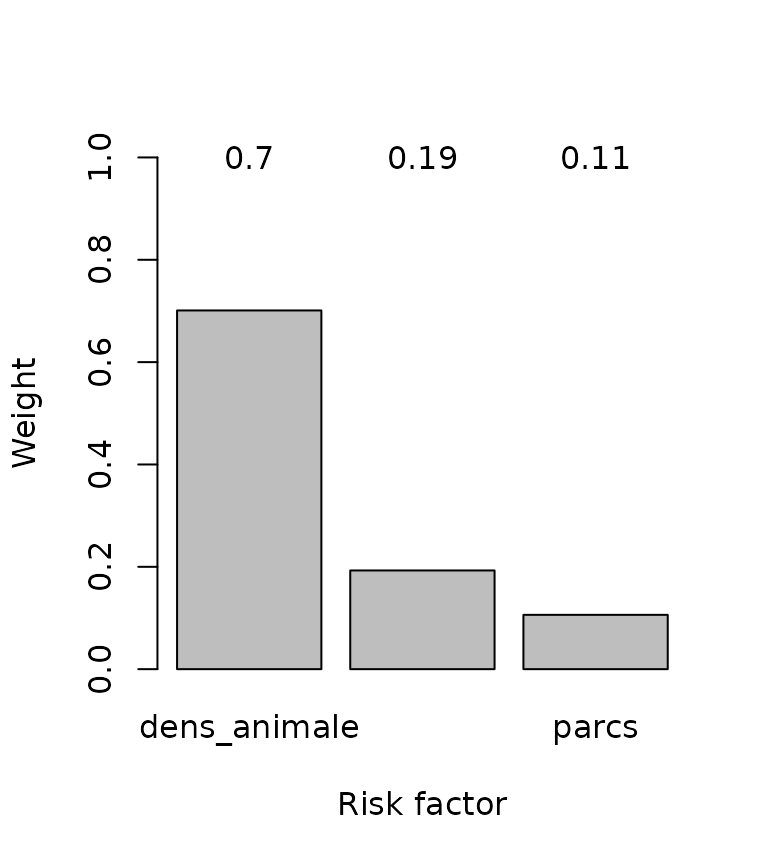
Pondération des facteurs de risque.
4. Calcul de la carte de risque
La fonction wlc() (pour weighted linear combination)
combine tous les facteurs de risque en utilisant les poids calculés
précedament, et produise une carte de risque qui couvre toute la
région.
risque_combine <- wlc(risques, w)
plot(risque_combine, col = hcl.colors(50))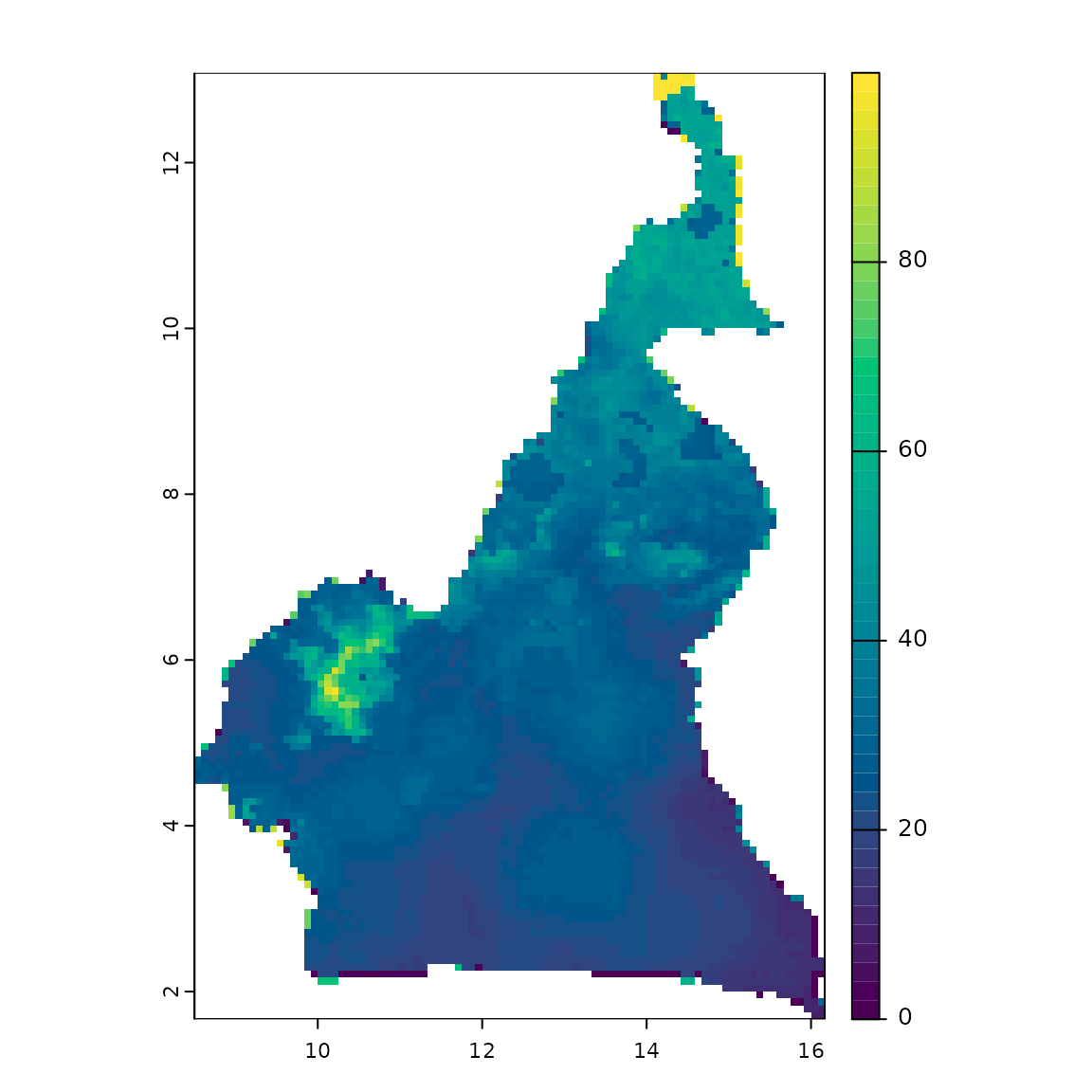
Carte de risque combiné.
Enfin, on peut produîre una carte par unité épidémiologique et dans une échelle de quelques niveaux de risque.
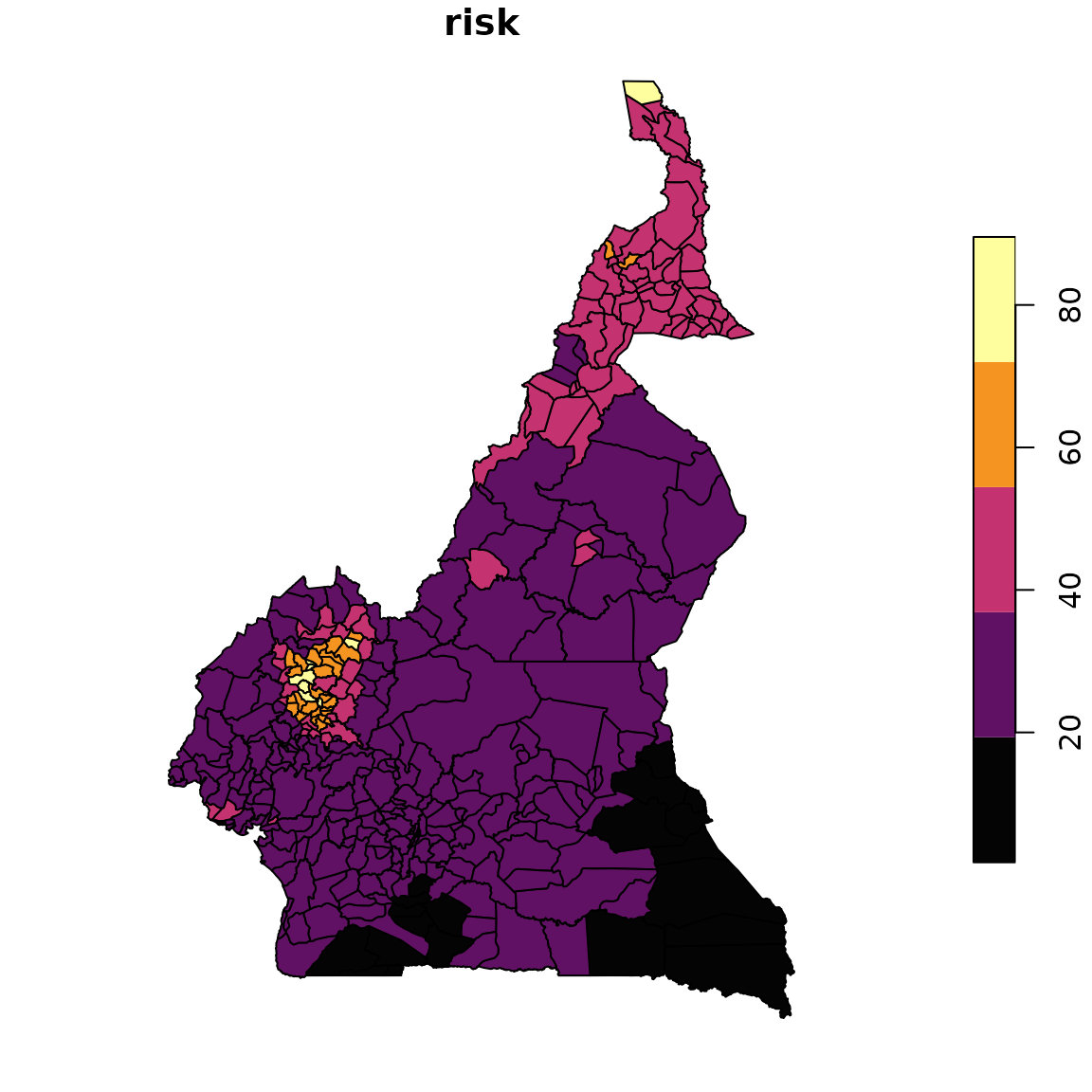
Carte de niveaux de risque par unité épidémiologique.